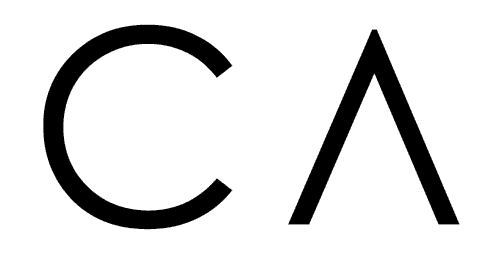Ajuster un modèle aux données#
Nous avons vu jusqu’ici plusieurs exemples de modèles en physique, et leur représentation numérique. Ces modèles sont construits pour représenter des données expérimentales ou des observations.
Les modèles dépendent d’un certain nombre de paramètres qu’il faut choisir afin de représenter au mieux les données. Une méthode possible est d’utiliser les données expérimentales ou les observations afin de trouver le meilleur jeu de paramètres qui les représente à l’aide d’un modèle donné. On dit que l’on fait un ajustement du modèle aux données. En anglais on appelle cela un fit, terme qui est souvent repris par les physiciens, même en français.
Une méthode pour trouver le meilleur ajustement d’un modèle aux données est la méthode des moindres carrés, ce que nous allons voir ici.
Paramètres d’un modèle#
Reprenons le modèle de l’accéleration constante vu dans la séquence sur la démarche de modélisation. Nous avons mesuré la vitesse \(v_i\) d’un objet pour des temps \(t_i\). L’objet est lâché avec une vitesse initiale vers le bas d’environ 1,6 m/s. Voici le résultat des mesures :
\(i\) |
0 |
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
|---|---|---|---|---|---|---|---|---|---|---|---|
\(t_i\) |
0.0 |
0.1 |
0.2 |
0.3 |
0.4 |
0.5 |
0.6 |
0.7 |
0.8 |
0.9 |
1.0 |
\(v_i\) |
0.338 |
-1.509 |
-5.301 |
-4.404 |
-6.967 |
-6.229 |
-5.279 |
-9.266 |
-8.847 |
-11.260 |
-12.040 |
Le modèle est :
Nous déclarons que \(v_0\) et \(g\) sont les paramètres du modèle. Il est d’usage en physique de noter les paramètres après un point-virgule dans la fonction : \(v(t;v_0,g)\), pour les distinguer de la variable libre \(t\).
Nous supposons que les paramètres du modèle, \(v_0\) et \(g\), ne sont pas connus a priori. Nous allons utiliser les données expérimentales pour trouver les meilleures valeurs possibles des paramètres, d’après ces données.
Représentons graphiquement les données et le modèle :
# Importation des modules
import numpy as np
import matplotlib.pyplot as plt
# Données experimentales
N = 11
t_exp = np.linspace(0, 1, N)
v_exp = np.array([0.338, -1.509, -5.301, -4.404, -6.967, -6.229, -5.279, -9.266, -8.847, -11.260, -12.040])
# Paramètres du modèle
g = 5
v0 = 0
# Discrétisation du modèle
t_mod = np.linspace(0, 1, N)
v_mod = v0 - g * t_exp
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
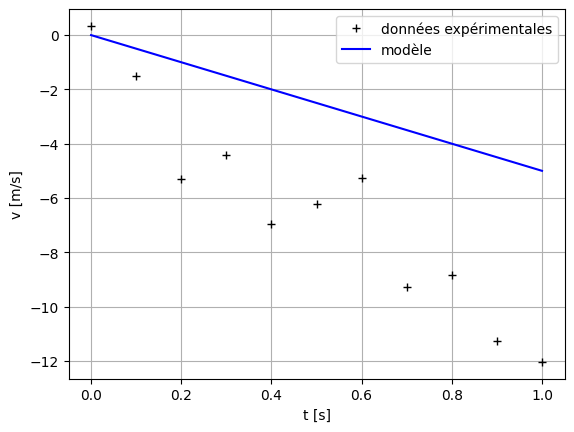
Exercice#
Ajuster la valeur des paramètres \(v_0\) et \(g\) à la main, c’est-à-dire en changeant directement leur valeur dans le script ci-dessus, afin que le modèle représente au mieux les données.
Quel est le critère qui vous permet de dire que le modèle représente mieux les données ?
# Paramètres du modèle
g = 10
v0 = -1
# Discrétisation du modèle
t_mod = np.linspace(0, 1, N)
v_mod = v0 - g * t_exp
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
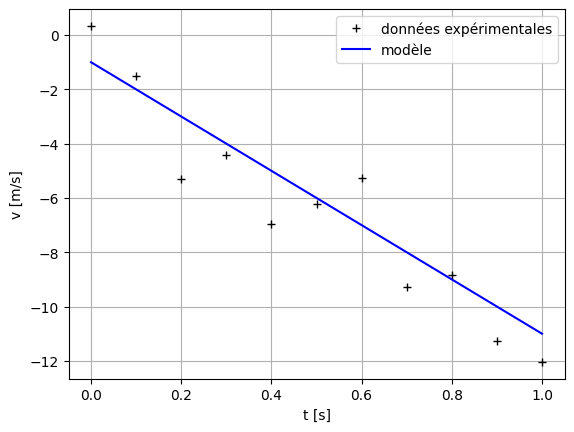
La méthode des moindres-carrés#
La méthode des moindres carrés permet d’utiliser un critère quantitatif afin de trouver le meilleur ajustement d’un modèle d’après des données expérimentales. Calculons la somme quadratique des écarts entre le modèle et les données du problème :
Les couples \((x_i,y_i)\) sont les données expérimentales, alors que \(y=P(x)\) est le modèle. Le but pour trouver le modèle le mieux ajusté aux données expérimentales est de minimiser la somme \(S\). On minimise les carrés des écarts, d’où le nom de la méthode des « moindres carrés ».
Si on applique cela au problème de l’accélération constante, on a :
\(S\) est une fonction des paramètres du problème, qui doit avoir un minimum pour le meilleur ajustement du modèle aux données (best fit en anglais).
Représentons graphiquement cette somme :
# Paramètres du modèle
g = 5
v0 = 0
# Discrétisation du modèle
t_mod = np.linspace(0, 1, N)
v_mod = v0 - g * t_exp
print(v_mod)
# Calcul de la somme S
Si = (v_exp - v_mod) ** 2
S = np.sum(Si)
print(Si.shape, S.shape)
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
# représentation des écarts entre le modèle et les données
for i in range(N - 1):
plt.plot((t_exp[i], t_exp[i]), (v_exp[i], v_mod[i]), '-r')
plt.title('S = ' + str(np.around(S,2)))
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
[ 0. -0.5 -1. -1.5 -2. -2.5 -3. -3.5 -4. -4.5 -5. ]
(11,) ()
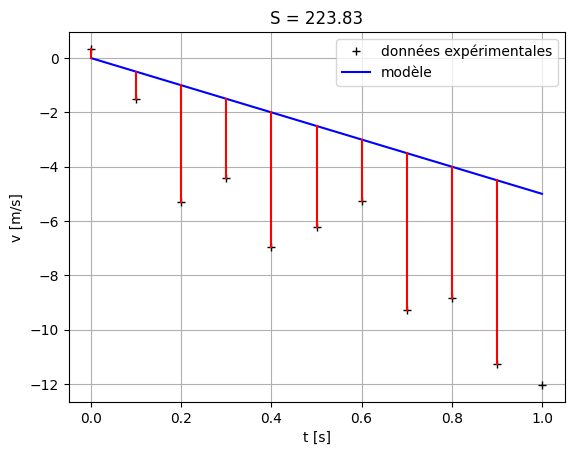
Exercice#
Dans la cellule ci-dessus, calculer \(S\) pour les paramètres trouvés à l’exercice précedent. Pouvez-vous améliorer la valeur des paramètres en vous basant sur la valeur de \(S\) ?
Pourquoi les éléments de la somme des écarts entre les données et le modèle sont-ils mis au carré ?
# Paramètres du modèle
g = 10
v0 = -1
# Discrétisation du modèle
t_mod = np.linspace(0, 1, N)
v_mod = v0 - g * t_exp
print(v_mod)
# Calcul de la somme S
Si = (v_exp - v_mod) ** 2
S = np.sum(Si)
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
# représentation des écarts entre le modèle et les données
for i in range(N - 1):
plt.plot((t_exp[i], t_exp[i]), (v_exp[i], v_mod[i]), '-r')
plt.title('S = ' + str(np.around(S,2)))
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
[ -1. -2. -3. -4. -5. -6. -7. -8. -9. -10. -11.]
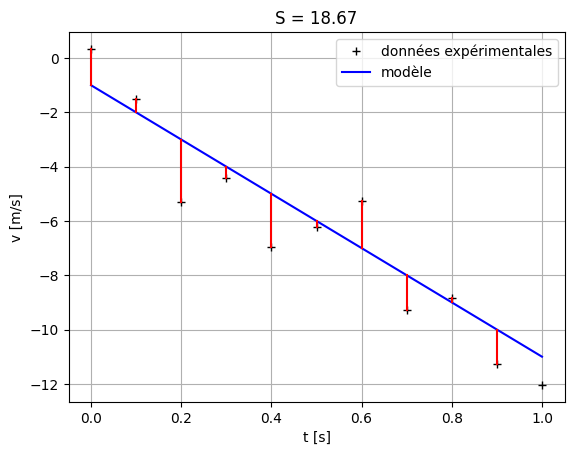
Régression linéaire#
Pour un modèle linéaire tel que celui de l’accélération constante, il est possible de trouver le minimum de la fonction \(S\) de façon analytique.
Le modèle linéaire est de la forme \(P(x; a, b) = a + b x\). La somme \(S\) s’écrit alors
Le polynôme \(S\) obtenu est du second degré en \(a\) et \(b\). Il peut s’écrire
Une condition nécessaire pour que \(S\) ait un minimum en \((a_m,b_m)\) est que le gradient de \(S\) s’annule au point \((a_m,b_m)\) :
\begin{eqnarray*} \frac{\partial S}{\partial a} (a_m,b_m) & = & 0 ;;;; \Rightarrow ;;;; N a_m + b_m \sum_{i=0}^{N-1} x_i = \sum_{i=0}^{N-1} y_i\ \frac{\partial S}{\partial b} (a_m,b_m) & = & 0 ;;;; \Rightarrow ;;;; a_m \sum_{i=0}^{N-1} x_i + b_m \sum_{i=0}^{N-1} x_i^2 = \sum_{i=0}^{N-1} x_i y_i \end{eqnarray*}
On obtient alors :
\begin{eqnarray*} a_m &=& \bar{y}-b_m \bar{x}\ b_m &=& \frac{\mathrm{cov}(x,y)}{\mathrm{var}(x)} \end{eqnarray*}
où
\begin{eqnarray*} & \bar{x} = \frac{1}{N} \sum_{i=0}^{N-1} x_{i} \ & \bar{y} = \frac{1}{N} \sum_{i=0}^{N-1} y_{i} \ & \mathrm{cov}(x,y) = \frac{1}{N} \sum_{i=0}^{N-1} (x_{i} - \bar{x}) (y_{i} - \bar{y_i}) = \left( \frac{1}{N} \sum_{i=0}^{N-1} x_{i} y_{i} \right) - \bar{x}\bar{y} \ & \mathrm{var}(x) = \frac{1}{N} \sum_{i=0}^{N-1} (x_{i} - \bar{x})^2 = \left( \frac{1}{N} \sum_{i=0}^{N-1} x_{i}^2 \right) - \bar{x}^2 \end{eqnarray*}
La fonction suivante renvoie les paramètres \(a\) et \(b\) de la régression linéaire pour un jeu de données expérimentales :
# Fonction régression linéaire
def reglin(xi, yi):
'''
Calcule les paramètres a et b du meilleur ajustement d'un modèle linéaire
y = ax + b sur un jeu de données (xi, yi)
'''
N = np.size(xi)
xm = np.mean(xi)
ym = np.mean(yi)
cov = 1 / N * np.sum(xi * yi) - xm * ym
var = 1 / N * np.sum(xi ** 2) - xm ** 2
b = cov / var
a = ym - b * xm
return (a, b)
Exercice#
Faire une régression linéaire sur les données temps, vitesse : \((t_{\mathrm{exp}},v_{\mathrm{exp}})\) en utilisant la fonction
reglin().Comparer le résultat obtenu avec les valeurs des paramètres trouvés « à la main » dans l’exercice précédent.
Calculer \(S\) et le comparer à celui obtenu à l’exercice précédent
Tracer les données et le modèle trouvé avec la régression linéaire
# 1
regl = reglin(t_exp, v_exp)
print(regl)
(np.float64(-0.9981818181818154), np.float64(-10.869818181818184))
# 2
print("La différence pour les valuers de g =", str(-g), str(regl[1]))
print("La différence pour les valuers de v_0 =" , str(v0), str(regl[0]))
La différence pour les valuers de g = -10 -10.869818181818184
La différence pour les valuers de v_0 = -1 -0.9981818181818154
# Paramètres du modèle
g = regl[1]
v0 = regl[0]
# Discrétisation du modèle
t_mod = np.linspace(0, 1, N)
v_mod = v0 + g * t_exp
print(v_mod)
# Calcul de la somme S
Si = (v_exp - v_mod) ** 2
S = np.sum(Si)
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
# représentation des écarts entre le modèle et les données
for i in range(N - 1):
plt.plot((t_exp[i], t_exp[i]), (v_exp[i], v_mod[i]), '-r')
plt.title('S = ' + str(np.around(S,2)))
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
[ -0.99818182 -2.08516364 -3.17214545 -4.25912727 -5.34610909
-6.43309091 -7.52007273 -8.60705455 -9.69403636 -10.78101818
-11.868 ]
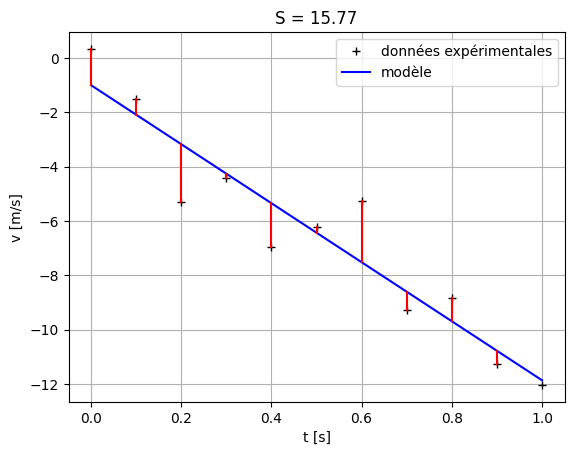
La fonction curve_fit#
La fonction curve_fit du module optimize de scipy permet d’ajuster n’importe quel modèle à des données expérimentales.
Voyons comment elle fonctionne avec les données de vitesse. Il faut d’abord définir la fonction python qui représente le modèle :
def modlin(x, a, b):
'''
Fonction qui représente le modèle linéaire
y(x; a, b) = a + bx
'''
return a + b * x
Attention
Dans la fonction python qui représente le modèle, il faut absolument que la variable libre soit le premier argument de la fonction. Suivent ensuite les paramètres du problème.
Nous importons ensuite la fonction curve_fit :
from scipy.optimize import curve_fit
---------------------------------------------------------------------------
ModuleNotFoundError Traceback (most recent call last)
Cell In[10], line 1
----> 1 from scipy.optimize import curve_fit
ModuleNotFoundError: No module named 'scipy'
Les arguments de la fonction curve_fit sont :
La fonction python qui représente le modèle
Le tableau qui représente les données \(x_i\)
Le tableau qui représente les données \(y_i\)
# Ajustement du modèle linéaire
solution = curve_fit(modlin, t_exp, v_exp, p0 = [0., 10.])
# Identification des paramètres du modèle
a, b = solution[0]
v0 = a
g = -b
# Affichage du résultat
print('v0 = {:4.2f} m.s**-1'.format(v0))
print('g = {:4.2f} m.s**-2'.format(g))
# Calcul du modèle
t_mod = np.linspace(0, 1, N)
v_mod = modlin(t_mod, a, b)
# Calcul de la somme S
Si = (v_exp - v_mod) ** 2
S = np.sum(Si)
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
plt.title('S = ' + str(S))
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
v0 = -1.00 m.s**-1
g = 10.87 m.s**-2
On voit que l’on obtient le même résultat qu’avec la régression linéaire. Ce ne sont pourtant pas les mêmes méthodes numériques qui sont employées. En augmentant le nombre de chiffres après la virgule utilisés pour afficher les paramètres trouvés avec chaque méthode, vous verrez que les résultats finissent par différer. On appelle cela la précision numérique de la méthode numérique. On le voit aussi sur les sommes \(S\) qui sont différentes, certes pas de beaucoup !
Attention
La fonction curve_fit() utilise des valeurs de départ par défaut égales à 1 pour tous les paramètres, avant d’ajuster ces valeurs grâce à un algorithme que l’on ne verra pas dans ce cours. Il arrive parfois que la fonction curve_fit() ne trouve pas de solution car les valeurs de départ des paramètres sont trop éloignées de la solution. Il faut alors utiliser l’option p0 de la fonction curve_fit(), où il faut spécifier le tableau des valeurs de départ voulues, qui soient plus approchées de la solution. Afin de connaître une valeur approximative de la solution, on peut comme dans le tout 1er exercice ajuster à la main les paramètres.
help(curve_fit)
Help on function curve_fit in module scipy.optimize.minpack:
curve_fit(f, xdata, ydata, p0=None, sigma=None, absolute_sigma=False, check_finite=True, bounds=(-inf, inf), method=None, jac=None, **kwargs)
Use non-linear least squares to fit a function, f, to data.
Assumes ``ydata = f(xdata, *params) + eps``
Parameters
----------
f : callable
The model function, f(x, ...). It must take the independent
variable as the first argument and the parameters to fit as
separate remaining arguments.
xdata : array_like or object
The independent variable where the data is measured.
Should usually be an M-length sequence or an (k,M)-shaped array for
functions with k predictors, but can actually be any object.
ydata : array_like
The dependent data, a length M array - nominally ``f(xdata, ...)``.
p0 : array_like, optional
Initial guess for the parameters (length N). If None, then the
initial values will all be 1 (if the number of parameters for the
function can be determined using introspection, otherwise a
ValueError is raised).
sigma : None or M-length sequence or MxM array, optional
Determines the uncertainty in `ydata`. If we define residuals as
``r = ydata - f(xdata, *popt)``, then the interpretation of `sigma`
depends on its number of dimensions:
- A 1-d `sigma` should contain values of standard deviations of
errors in `ydata`. In this case, the optimized function is
``chisq = sum((r / sigma) ** 2)``.
- A 2-d `sigma` should contain the covariance matrix of
errors in `ydata`. In this case, the optimized function is
``chisq = r.T @ inv(sigma) @ r``.
.. versionadded:: 0.19
None (default) is equivalent of 1-d `sigma` filled with ones.
absolute_sigma : bool, optional
If True, `sigma` is used in an absolute sense and the estimated parameter
covariance `pcov` reflects these absolute values.
If False, only the relative magnitudes of the `sigma` values matter.
The returned parameter covariance matrix `pcov` is based on scaling
`sigma` by a constant factor. This constant is set by demanding that the
reduced `chisq` for the optimal parameters `popt` when using the
*scaled* `sigma` equals unity. In other words, `sigma` is scaled to
match the sample variance of the residuals after the fit.
Mathematically,
``pcov(absolute_sigma=False) = pcov(absolute_sigma=True) * chisq(popt)/(M-N)``
check_finite : bool, optional
If True, check that the input arrays do not contain nans of infs,
and raise a ValueError if they do. Setting this parameter to
False may silently produce nonsensical results if the input arrays
do contain nans. Default is True.
bounds : 2-tuple of array_like, optional
Lower and upper bounds on parameters. Defaults to no bounds.
Each element of the tuple must be either an array with the length equal
to the number of parameters, or a scalar (in which case the bound is
taken to be the same for all parameters.) Use ``np.inf`` with an
appropriate sign to disable bounds on all or some parameters.
.. versionadded:: 0.17
method : {'lm', 'trf', 'dogbox'}, optional
Method to use for optimization. See `least_squares` for more details.
Default is 'lm' for unconstrained problems and 'trf' if `bounds` are
provided. The method 'lm' won't work when the number of observations
is less than the number of variables, use 'trf' or 'dogbox' in this
case.
.. versionadded:: 0.17
jac : callable, string or None, optional
Function with signature ``jac(x, ...)`` which computes the Jacobian
matrix of the model function with respect to parameters as a dense
array_like structure. It will be scaled according to provided `sigma`.
If None (default), the Jacobian will be estimated numerically.
String keywords for 'trf' and 'dogbox' methods can be used to select
a finite difference scheme, see `least_squares`.
.. versionadded:: 0.18
kwargs
Keyword arguments passed to `leastsq` for ``method='lm'`` or
`least_squares` otherwise.
Returns
-------
popt : array
Optimal values for the parameters so that the sum of the squared
residuals of ``f(xdata, *popt) - ydata`` is minimized
pcov : 2d array
The estimated covariance of popt. The diagonals provide the variance
of the parameter estimate. To compute one standard deviation errors
on the parameters use ``perr = np.sqrt(np.diag(pcov))``.
How the `sigma` parameter affects the estimated covariance
depends on `absolute_sigma` argument, as described above.
If the Jacobian matrix at the solution doesn't have a full rank, then
'lm' method returns a matrix filled with ``np.inf``, on the other hand
'trf' and 'dogbox' methods use Moore-Penrose pseudoinverse to compute
the covariance matrix.
Raises
------
ValueError
if either `ydata` or `xdata` contain NaNs, or if incompatible options
are used.
RuntimeError
if the least-squares minimization fails.
OptimizeWarning
if covariance of the parameters can not be estimated.
See Also
--------
least_squares : Minimize the sum of squares of nonlinear functions.
scipy.stats.linregress : Calculate a linear least squares regression for
two sets of measurements.
Notes
-----
With ``method='lm'``, the algorithm uses the Levenberg-Marquardt algorithm
through `leastsq`. Note that this algorithm can only deal with
unconstrained problems.
Box constraints can be handled by methods 'trf' and 'dogbox'. Refer to
the docstring of `least_squares` for more information.
Examples
--------
>>> import matplotlib.pyplot as plt
>>> from scipy.optimize import curve_fit
>>> def func(x, a, b, c):
... return a * np.exp(-b * x) + c
Define the data to be fit with some noise:
>>> xdata = np.linspace(0, 4, 50)
>>> y = func(xdata, 2.5, 1.3, 0.5)
>>> np.random.seed(1729)
>>> y_noise = 0.2 * np.random.normal(size=xdata.size)
>>> ydata = y + y_noise
>>> plt.plot(xdata, ydata, 'b-', label='data')
Fit for the parameters a, b, c of the function `func`:
>>> popt, pcov = curve_fit(func, xdata, ydata)
>>> popt
array([ 2.55423706, 1.35190947, 0.47450618])
>>> plt.plot(xdata, func(xdata, *popt), 'r-',
... label='fit: a=%5.3f, b=%5.3f, c=%5.3f' % tuple(popt))
Constrain the optimization to the region of ``0 <= a <= 3``,
``0 <= b <= 1`` and ``0 <= c <= 0.5``:
>>> popt, pcov = curve_fit(func, xdata, ydata, bounds=(0, [3., 1., 0.5]))
>>> popt
array([ 2.43708906, 1. , 0.35015434])
>>> plt.plot(xdata, func(xdata, *popt), 'g--',
... label='fit: a=%5.3f, b=%5.3f, c=%5.3f' % tuple(popt))
>>> plt.xlabel('x')
>>> plt.ylabel('y')
>>> plt.legend()
>>> plt.show()
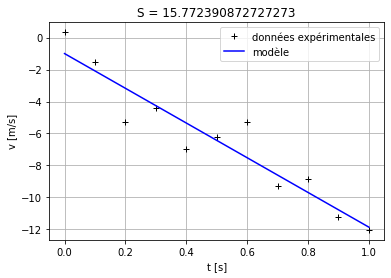
Exercice#
Grâce à la fonction curve_fit, on peut facilement changer de modèle. Définir un modèle quadratique de la forme \(y(x;a,b,c) = a + bx + cx^2\), et ajuster les paramètres sur les données de vitesse. Calculer la somme \(S\) et représenter le modèle et les données.
def quadmodel(x, a, b, c):
return (a + (b * x)) + (c * (x ** 2))
# On peut définir une valeur de 'c' pour vérifier si çà marche la fonction 'quadmodel'
c = -3
v_mod = quadmodel(t_mod, a, b, c)
# Calcul de la somme S
Si = (v_exp - v_mod) ** 2
S = np.sum(Si)
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
plt.title('S = ' + str(S))
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
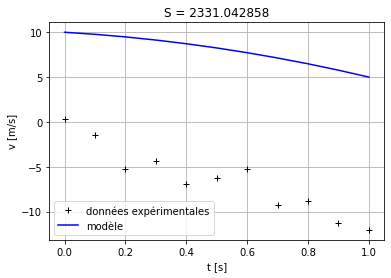
c = -2
y = quadmodel(t_mod, a, b, c)
np.random.seed(1729)
y_noise = 0.2 * np.random.normal(size=t_exp.size)
ydata = y + y_noise
solution, _ = curve_fit(quadmodel, t_exp,ydata)
# Identification des paramètres du modèle
a, b, c2 = solution
v0 = a
g = -b
# Affichage du résultat
print('v0 = {:4.2f} m.s**-1'.format(v0))
print('g = {:4.2f} m.s**-2'.format(g))
print('c = {:4.2f}'.format(c2))
# Calcul du modèle
t_mod = np.linspace(0, 1, N)
v_mod = quadmodel(t_mod, a, b, c2)
# Calcul de la somme S
Si = (v_exp - v_mod) ** 2
S = np.sum(Si)
# Représentation graphique du modèle et des données
plt.plot(t_exp, v_exp, '+k', label = 'données expérimentales')
plt.plot(t_mod, v_mod, '-b', label = 'modèle')
plt.title('S = ' + str(S))
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.legend()
plt.show()
v0 = -1.29 m.s**-1
g = 9.10 m.s**-2
c = -2.32
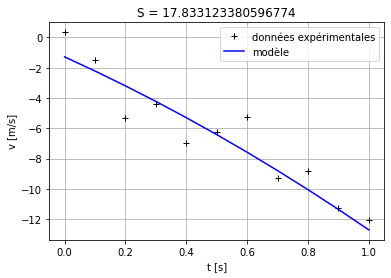
Erreurs de mesures#
Vous savez pour avoir déjà fait des expériences de physique en Travaux Pratiques, que l’on attache toujours une erreur estimée aux mesures expérimentales. Il est possible avec la fonction errorbar() de matplotlib d’afficher à la fois les mesures et leur barre d’erreur.
Dans notre exemple, on suppose que chaque mesure a la même erreur de mesure qui est de \(\pm 2\) m.s \(^{-1}\). On peut alors représenter les barres d’erreur avec l’option yerr (par défaut le 3ème argument) de la fonction errorbar(), qui est un tableau de même dimension que les données contenant les erreurs sur chaque point de mesure:
# définition du tableau des erreurs de mesure
erreurs = 2. * np.ones(v_exp.shape) # m/s
# Représentation graphique des données avec les barres d'erreur
plt.errorbar(t_exp, v_exp, yerr=erreurs, marker = '+', linestyle = '')
# Option du graphique
plt.xlabel('t [s]')
plt.ylabel('v [m/s]')
plt.grid()
plt.show()
Erreurs sur les paramètres ajustés#
Nous ne rentrerons pas dans les détails, mais la fonction curve_fit() permet d’estimer l’erreur sur les paramètres du modèle ajustés aux données grâce à la donnée des erreurs de mesure. Il faut alors spécifier les erreurs de mesure grâce à l’option sigma de la fonction curve_fit(), avec le tableau contenant les erreurs \(\sigma_{y_i}\) sur chaque mesure.
La somme quadratique des écarts entre le modèle et les données du problème est alors pondérée par les incertitudes de mesure :
Ainsi, les mesures avec une grande incertitude vont moins contribuer à la somme, et donc auront moins d’impact sur l’ajustement du modèle aux données.
On pourra remarquer qu’en plus du tableau contenant les paramètres ajustés, un deuxième tableau est retourné par la fonction curve_fit(). Il s’agit de la matrice de covariance de ces paramètres (que l’on notera pcov dans le code ci-dessous). On peut extraire de la matrice de covariance l’incertitude sur chacun des paramètres, donnée par la racine carrée des éléments diagonaux : perr = np.sqrt(np.diag(pcov)).
Par exemple, estimons l’erreur sur les paramètres ajustés pour le modèle linéaire :
# Ajustement du modèle linéaire
solution, pcov = curve_fit(modlin, t_exp, v_exp, sigma = erreurs, absolute_sigma = True)
# Identification des paramètres du modèle
a, b = solution
v0 = a
g = -b
# Calcul de l'incertitude sur les paramètres ajustés
perr = np.sqrt(np.diag(pcov))
# Affichage
print('v0 = %5.3f \u00B1 %5.3f m/s' % (v0, perr[0]))
print('g = %5.3f \u00B1 %5.3f m/s' % (g, perr[1]))
v0 = -0.998 ± 1.128 m/s
g = 10.870 ± 1.907 m/s
Attention
Par défaut la fonction curve_fit applique un facteur multiplicatif global sur les erreurs afin de normaliser la somme \(S\), c’est-à-dire pour que \(S=1\). Ainsi, par défaut, les erreurs n’ont pas un sens absolu mais seulement relatif entre elles. Afin d’obtenir l’erreur absolue, il faut mettre l’option absolute_sigma = True dans la fonction curve_fit().